研究miRNA,这些数据库你必须得知道!| 常用数据库汇总
- 摘要:
- (来源:科研小助手公众号;公众号ID:SciRes) miRNA是近年来在多种真核细胞及病毒中发现的一类来源内源性染色体上的非编码单链RNA,长度为21~25nt的短序列,在进化上具有高度的保守性,能够通过与靶mRNA特异性的碱基互补配对,引起靶mRNA降解或者抑制其翻译,从而对基因进行转录后的表达调控。
miRNA是近年来在多种真核细胞及病毒中发现的一类来源内源性染色体上的非编码单链RNA,长度为21~25nt的短序列,在进化上具有高度的保守性,能够通过与靶mRNA特异性的碱基互补配对,引起靶mRNA降解或者抑制其翻译,从而对基因进行转录后的表达调控。miRNA由一段具有发夹环结构的长度为70~80个核苷酸的miRNA前体(pre-miRNA)剪切后生成。它通过与其目标mRNA分子的3′端非编码区域(3-untranslated region, 3′ UTR)互补导致该mRNA分子的翻译受到抑制。最先发现的miRNAs是线虫中控制发育时序的lin-4和let-7基因。现已发现miRNA广泛地存在哺乳动物、线虫、果蝇和植物等生物中。除了lin-4和let-7基因外,其他miRNAs现在统一用miR-#表示miRNA,同一物种内相同或极相近似的miRNA可以使用相同的数字,只是进一步在数字之后加数字或字母作为后缀以区别其基因在序列上只有微小的差别。尽管miRNA基因不编码蛋白质,但其编码的RNA在生物的整个生命过程中发挥着重要作用。

miRBase
http://www.mirbase.org

miRBase数据库是一个提供包括已发表的miRNA序列数据、注释、预测基因靶标等信息的全方位数据库,是存储miRNA信息最主要的公共数据库之一。该数据库于2014年6月更新为最新版本V21.0,包含223个物种的35828个成熟的miRNA序列。该数据库提供便捷的网上查询服务,允许用户使用关键词或序列在线搜索已知的miRNA和靶标信息(仅包含已有的靶标信息,所以会出现部分miRNA靶标信息无的现象)。该数据库用于miRNA信息查询较多,靶关系预测较少。
TargetScan
http://www.targetscan.org
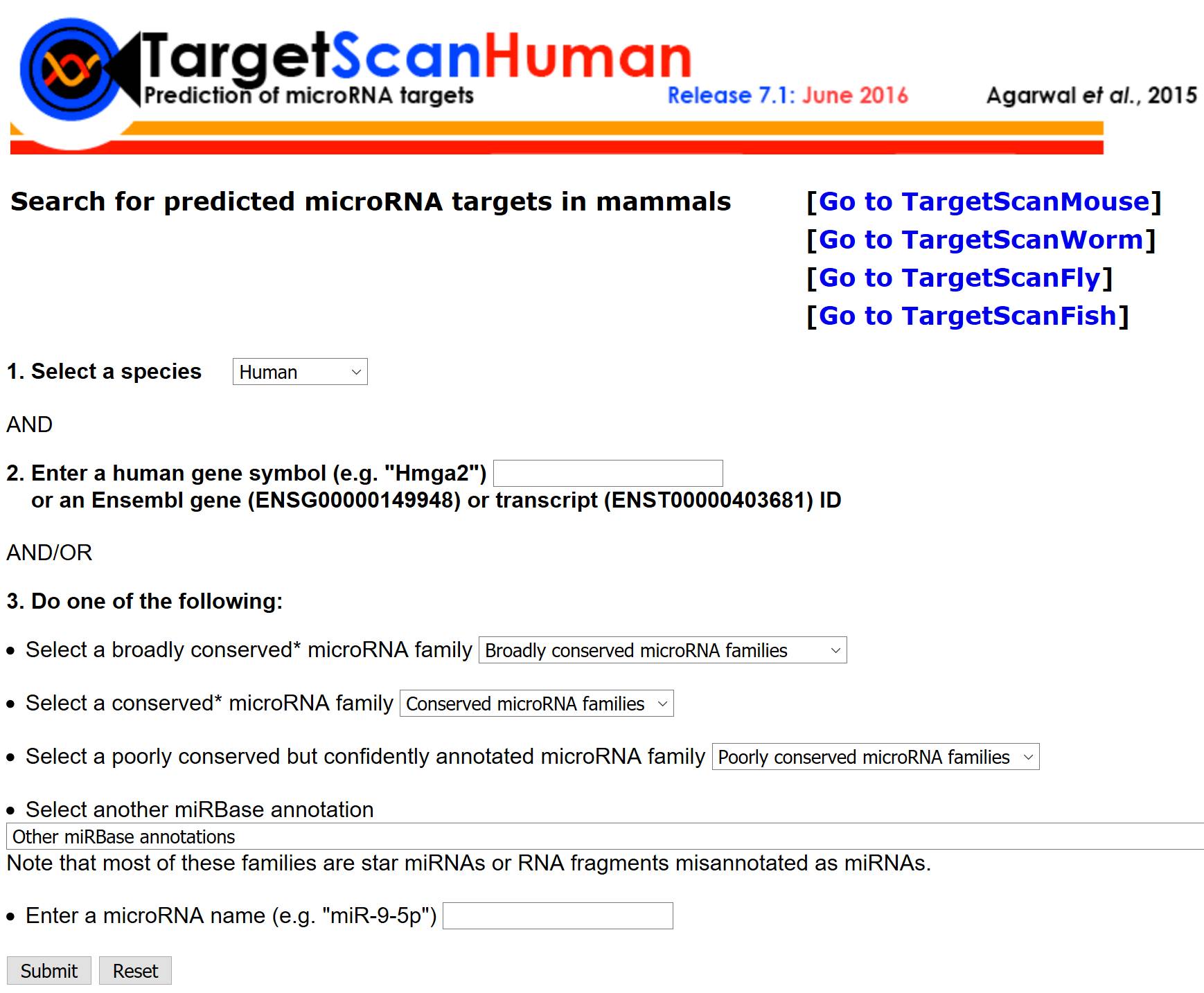
TargetScan数据库是大家比较常用的预测miRNA靶基因数据库,主要通过搜索和每条miRNA种子区域匹配的保守的8mer和7mer位点来预测靶基因。该数据库提供人、小鼠、大鼠、奶牛、狗、猩猩、恒河猴、负鼠、鸡和青蛙等动物信息。最新更新时间为2015年8月,版本为V7.0。
microRNA.org
http://www.microrna.org/
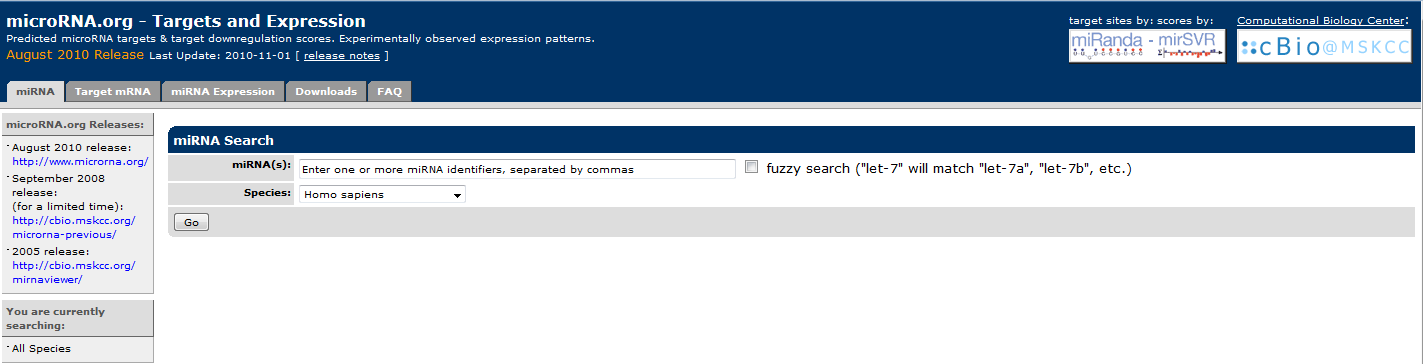
该数据库提供了关于人类、小鼠、大鼠、果蝇和斑马鱼基因组的microRNA靶目标的预测信息以及miRNA在不同组织的表达谱,支持通过miRNA预测靶基因,也支持通过mRNA分析相关的miRNA。最新更新时间为2010年,好久不更新啦~
starBase v2.0
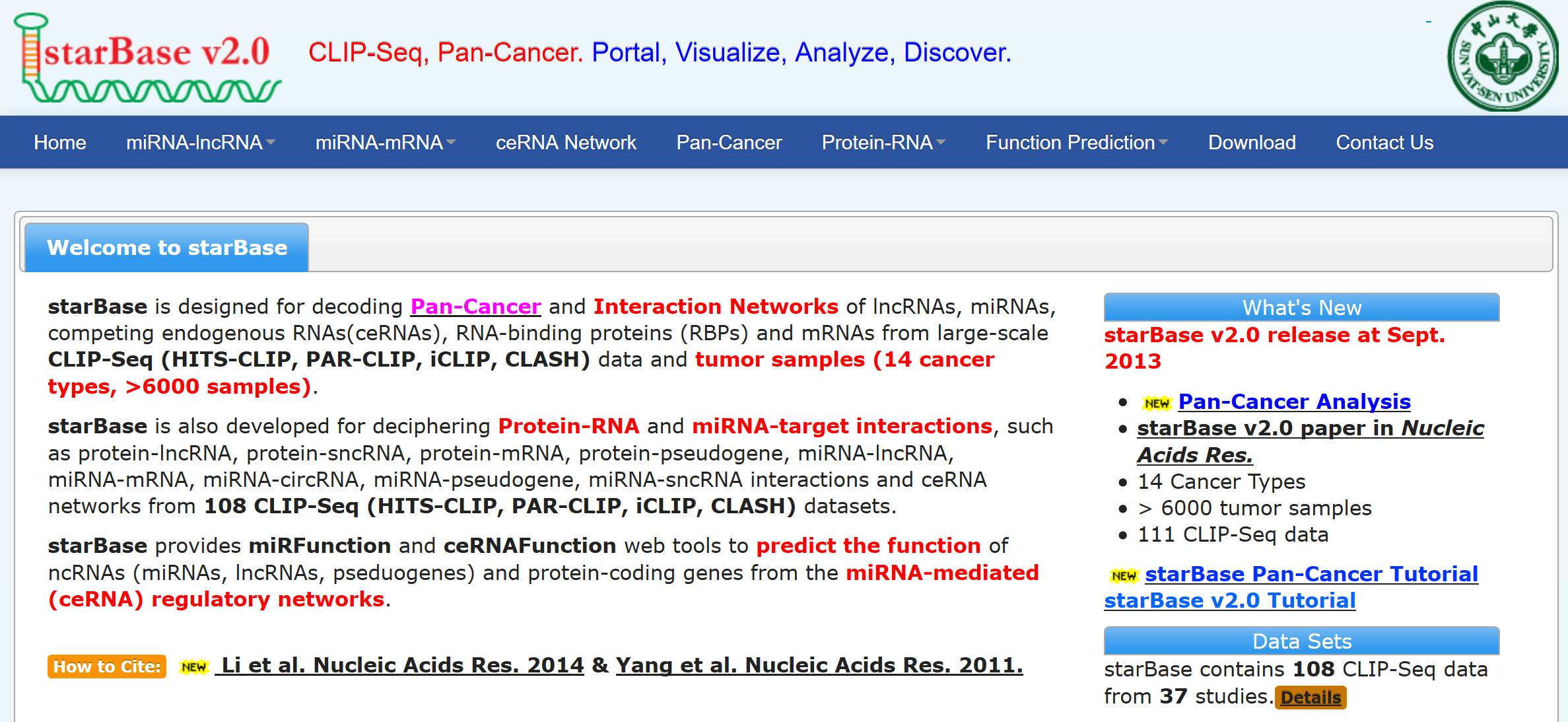
该数据库采集了6000多份样本,14种癌症来自于37个独立研究的108份CLIP-seq数据,同时辅助降解组实验数据搜寻miRNA的靶标,提供了各式各样的可视化界面去探讨miRNA靶标。除了miRNA和靶标mRNA之间关系,该数据库还进行lncRNA、circRNA、protein与mRNA之间的相互作用分析,并分析了ceRNA机制。该数据库主要包含人、小鼠、线虫3个物种信息。
PITA
http://genie.weizmann.ac.il/pubs/mir07/mir07_dyn_data.html

该数据库基于靶位点的可接性(target-site accessibility)和自由能预测miRNA 的靶标,是著名的生物信息学家Segal实验室开发的。主要包含human、mouse、fly和worm的信息,使用者可以通过miRNA预测靶基因,也可以通过mRNA预测miRNA信息,无论是miRNA还是mRNA均可通过提供name或ID进行分析。
PicTar
http://www.pictar.org/

PicTar是通过一定计算法则来鉴定microRNA的靶目标的。该可搜索的网站可以提供以下物种的关于microRNA靶目标的预测详细信息,包括:脊椎动物、七个果蝇种类、三个线虫种类和人类的非保守但共表达的microRNA的靶目标(例如:表达在同一组织的microRNA和mRNA)。
RNAhybrid
http://bibiserv.techfak.uni-bielefeld.de/rnahybrid/

RNAhybrid是Behmsmeier M等[15]基于miRNA和靶基因二聚体二级结构开发的miRNA靶基因预测软件。RNAhybrid预测算法禁止分子内、miRNA分子间及靶基因间形成二聚体,根据miRNA和靶基因间结合能探测最佳的靶位点。尽管随着靶基因序列长度增加,运算复杂度也相应增加,但RNAhybrid和其它RNA二级结构预测软件诸如mfold, RNAfold, RNAcofold和pairfold相比,仍具有明显的速度优势。此外,RNAhybrid允许用户自定义自由能阈值及p值,也允许用户设置杂交位点的偏向,如杂交位点必须包含miRNA 5’端2-7nt等。
miRWalk 2.0
http://zmf.umm.uni-heidelberg.de/apps/zmf/mirwalk2/
miRWalkis是一个综合性数据库,提供来自人类、小鼠和大鼠的miRNA的预测信息和经过验证的位于其靶基因上的结位点。
DIANA-microT
http://diana.cslab.ece.ntua.gr/microT/
DIANA-microT是KiriakidouM等基于实验和计算生物学方法开发的miRNA靶基因预测软件。和miRanda预测结果中可能出现一个miRNA对应多个靶位点或多个miRNA对应一个靶位点而丢掉了miRNA调控单个靶位点不同的是,DIANA-microT考虑了miRNA调控单个靶位点的情况。DIANA-microT预测算法基于以下两点来判别miRNA靶基因:①miRNA和靶基因间的高亲和力,主要通过结合能来衡量。②影响miRNA和靶基因所形成二聚体茎环结构环部位置和环大小的miRNA相关蛋白可能指导miRNA和靶基因的相互作用。
miRNAMap
http://mirnamap.mbc.nctu.edu.tw/
miRNAMap数据库搜集了多个物种的经过试验证明的microRNA和miRNA靶基因,其中包括人类的、小鼠的、大鼠的以及其他多细胞动物的基因组。
Deepbase v2.0
http://rna.sysu.edu.cn/deepBase/

DeepBase从新一代测序技术(深度测序技术,deep-sequencing)数据中注释和鉴定microRNA,piRNA,siRNA基因及长非编码RNA基因及其他们的表达模式。
除上述比较常用的数据库外,还有很多其他miRNA靶基因预测分析数据库,但这些数据库与以上所列数据库除了鉴定靶基因标准有些许差异外,其他方面大同小异,上述数据库均经过科研小助手亲自验证可用。研究miRNA的基本上掌握上述数据库就够用了!在本文中我们暂不就各个数据库的使用展开描述,后续我们会挑选几个具有代表性的详细讲解其使用,敬请期待!
·END·